Winzige "Blinker" für die superauflösende Mikroskopie
Forscher am Max-Planck-Institut für Biochemie verbessern die parallele Untersuchung mehrerer Strukturen mit superauflösender Mikroskopie.
- Mit fluoreszenten Sonden für die superauflösende Mikroskopie können nur wenige Strukturen gleichzeitig untersucht werden
- Unterschiedliche Blinkfrequenzen der Sonden werden eingesetzt um Ziele zu unterscheiden
- Die Methode erhöht die Anzahl der Strukturen, die parallel aufgenommen werden können
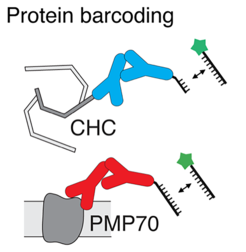
Blinksignale für die Nanowelt: Ralf Jungmann, Leiter der Forschungsgruppe Molekulare Bildgebung und Bionanotechnologie am Max-Planck-Institut für Biochemie (Martinsried) und Professor für Experimentalphysik an der LMU, nutzt winzige fluoreszierende Sonden, um Vorgänge auf molekularer Ebene sichtbar zu machen. Nun hat er mit seinem Team einen entscheidenden Fortschritt erzielt: Die Wissenschaftler entwickelten eine Methode, mit der sie zahlreiche verschiedene Moleküle in einer Zelle gleichzeitig erfassen können. Dies könnte zukünftig bessere Diagnosemöglichkeiten eröffnen. Über ihre Ergebnisse berichten die Wissenschaftler im Fachmagazin Nano Letters.
„Unser Konzept basiert auf der Bildgebungsmethode DNA-PAINT, bei der fluoreszierende Sonden auf der Basis synthetischer DNA verwendet werden, um Vorgänge auf molekularer Ebene sichtbar zu machen“, sagt Jungmann. Dabei sind Farbstoffe an kurze DNA-Stränge gebunden, die detektiert werden, wenn der DNA-Strang an sein Gegenstück bindet. Durch wiederholtes An- und Abbinden der Stränge entsteht dann ein Blinksignal. Bisher war diese Methode auf eine serielle Detektion ausgelegt, sodass pro Messdurchgang nur eine Molekülspezies abgebildet werden konnte.
In dem neuen Ansatz werden die Zielmoleküle so modifiziert, dass sie alle von derselben Sonde angesteuert werden – aber jeweils eine unterschiedliche Anzahl von Bindeplätzen für die Sonde aufweisen. „Durch die Modifikation wird das Blinksignal verändert: Wenn beispielsweise Molekül A fünf Bindestellen aufweist und Molekül B zehn, dann blinkt B doppelt so schnell wie A – auf diese Weise bekommt jedes Zielmolekül einen charakteristischen Blinzelcode“, sagt Jungmann. „Mithilfe dieser Methode können wir theoretisch hunderte molekulare Bestandteile wie Proteine und Nukleinsäuren in ungefähr 15 bis 20 Minuten mit hoher Genauigkeit auslesen.“ In einem ersten Test gelang es den Forschern, verschiedene RNA-Moleküle und Proteine gleichzeitig in der Zelle zu detektieren. „Insgesamt können wir derzeit maximal 124 Ziele gleichzeitig abbilden, die unterschiedlich blinken. Gezeigt haben wir das mit Hilfe sogenannter DNA-Origami-Strukturen“, sagt Jungmann. Der Ansatz eröffnet damit neue Möglichkeiten, das Protein- und RNA-Inventar der Zelle zu untersuchen und krankheitsbedingte Veränderungen oder therapeutisch relevante Strukturen wie etwa bestimmte Oberflächenproteine zu erfassen. [göd]
Originalpublikation:
O. Wade, J.B. Woehrstein, P.C. Nickels, S. Strauss, F. Steht, J. Stein, F. Schueder, M.T. Strauss, M. Ganji, J. Schnitzbauer, H. Grabmayr, P. Yin, P. Schwille, R. Jungmann. 124-Color Super-resolution Imaging by Engineering DNA-PAINT Blinking Kinetics. Nano Letters, März 2019
http://pubs.acs.org/doi/abs/10.1021/acs.nanolett.9b00508












