Neue Markierungssonden im Nanomaßstab
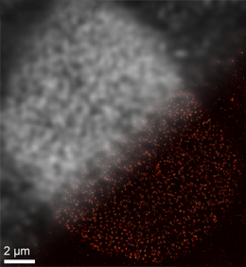
SOMAmer-basierte Markierungssonden für DNA-PAINT ermöglichen verbesserte Superauflösungsmikroskopie. Oben links: traditionelle beugungsbegrenzte Abbildung von Kernporen in der Kernhülle. Unten rechts: superaufgelöste DNA-PAINT-Aufnahme ermöglicht durch SOMAmer-Sonden.
Das Forschungsgebiet von Ralf Jungmann führt ihn in unvorstellbar kleine Sphären. Der Forschungsgruppenleiter für "Molekulare Bildgebung und Bionanotechnologie" an MPI für Biochemie und Professor für Experimentalphysik an der LMU entwickelt Mikroskopieverfahren, die molekulare Vorgänge auf zellulärer Ebene direkt sichtbar machen können. Dabei werden fluoreszierende Sonden aus kurzen DNA-Strängen an den Ort des Geschehens gebracht, die dort an Proteine binden und diese dadurch sozusagen von selbst zum Leuchten bringen. Diese Methode haben die Forscher DNA-PAINT getauft.
Eine der größten Herausforderungen für diese sogenannte superauflösende Fluoreszenzmikroskopie sind derzeit die Markierungssonden – sie sind schlicht nicht klein genug. „Wir arbeiten mit einer Ortsauflösung unter zehn Nanometern. Die herkömmlichen Markierungssonden sind dafür viel zu groß. Das hat bislang das ganze Forschungsfeld behindert“, beschreibt Ralf Jungmann den Ausgangspunkt seiner aktuell im Fachjournal Nature Methods veröffentlichten Studie. Darin hat das Team um Jungmann, der auch die Forschungsgruppe Molekulare Bildgebung und Bionanotechnologie am Max-Planck-Institut für Biochemie (Martinsried) leitet, eine neue Methode untersucht, Markierungssonden herzustellen: den Einsatz von sogenannten Aptameren, kleinen aus DNA bestehenden Molekülen, die aufgrund ihrer einzigartigen 3D-Struktur spezifisch an Proteine binden können.
„Die ideale Sonde, um Proteine effizient zu markieren, muss mehrere Voraussetzungen erfüllen“, sagt Sebastian Strauß aus der Arbeitsgruppe von Ralf Jungmann und Erstautor der Veröffentlichung: „Sie muss so klein wie möglich sein und auch zur quantitativen Färbung eingesetzt werden können. Außerdem sollte eine große Bibliothek dieser Sonden verfügbar sein, um möglichst viele unterschiedliche Zielproteine damit markieren zu können.“ Um das Potenzial von Aptameren zu untersuchen, haben die LMU-Physiker mit dem amerikanischen Unternehmen SomaLogic zusammengearbeitet, das für eine andere Anwendung bereits eine große Reihe an modifizierten Aptameren entwickelt hat, die an tausende verschiedene Proteine binden können. In der jetzigen Studie konnten die Forscher das Potenzial dieser modifizierten Aptamere als Markierungssonden am Beispiel von sieben unterschiedlichen Zielproteinen nachweisen.
„Wir gehen davon aus, dass die neue Methode das Feld der superauflösenden Mikroskopie besonders im Hinblick auf die Anwendbarkeit in der Biologie entscheidend voranbringen wird“, sagt Ralf Jungmann. Sein Ziel ist es, mithilfe von DNA-PAINT sehr viele Proteine und deren Interaktionen gleichzeitig zu beobachten. In kommenden Studien wird das Team daher mit der neuen Markierungsmethode ganze Proteinnetzwerke mit hoher Auflösung sichtbar machen. „Wir werden biologische und biomedizinische Fragestellungen angehen, die bislang nicht untersucht werden konnten.“
Originalpublikation
S. Strauss, P.C. Nickels, M.T. Strauss, V.J. Sabinina, J. Ellenberg, J.D. Carter, S. Gupta, N. Janjic & R. Jungmann: „Modified aptamers enable quantitative sub-10-nm cellular DNA-PAINT imaging“. Nature Methods, August 2018
DOI: https://doi.org/10.1038/s41592-018-0105-0












