Ein molekularer Aktenvernichter für RNA - Max-Planck-Forscher entschlüsseln Struktur der RNA-Abbau-Maschinerie
Ähnlich einem Aktenvernichter zum Zerkleinern von nicht mehr benötigten oder potenziell gefährlichen Dokumenten verwenden Zellen molekulare Maschinen, die überflüssige oder defekte Makromoleküle abbauen. Wissenschaftler am Max-Planck-Institut für Biochemie in Martinsried bei München haben jetzt die Struktur und Funktionsweise des Exosoms entschlüsselt, das Ribonukleinsäuren (RNA) in Eukaryoten abbaut. RNA-Moleküle liegen in allen Zellen in großer Menge vor und übernehmen dort vielfältige Aufgaben. Sie ermöglichen es zum Beispiel, die in den Genen gespeicherte Information in Proteine zu übersetzen. Die Ergebnisse der Forscher zeigen, dass die Struktur und die Funktionsweise des Exosoms in allen Lebensformen weitgehend gleich sind. Die Studie wurde jetzt im Journal Nature veröffentlicht.
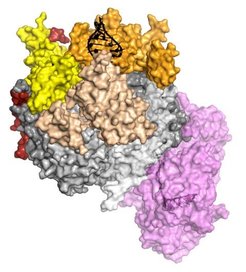
Wenn bei der Herstellung von RNA-Molekülen Fehler auftreten oder RNA sich unkontrolliert anhäuft, kann dies die Zelle schädigen. Deshalb ist die Beseitigung von defekter oder nicht mehr benötigter RNA ein wichtiger Schritt für den Stoffwechsel einer Zelle. Das Exosom zerschneidet als Multi-Proteinkomplex RNA in kleine Stücke und spielt damit eine Schlüsselrolle im Abbau-Prozess. Zusätzlich wandelt es bestimmte RNA-Moleküle in ihre reife Form um. Die molekularen Mechanismen, mit denen das Exosom all diese Funktionen erfüllen kann, waren bisher noch wenig verstanden.
Allgegenwärtiger molekularer Aktenvernichter
Debora Makino, Wissenschaftlerin in der Forschungsabteilung „Zelluläre Strukturbiologie“ um Elena Conti, hat jetzt auf atomarem Level ein Bild des kompletten Exosoms aus Eukaryoten erstellt - zusammen mit einem gebundenen RNA Molekül. Die Struktur dieses Komplexes ermöglicht es den Wissenschaftlern zu verstehen, wie das Exosom im Detail arbeitet.
„Es handelt sich um eine sehr aufwändige molekulare Maschine: der Exosom-Komplex bildet ein hohles Fass aus neun unterschiedlichen Proteinen. Durch einen Kanal in seinem Inneren werden die RNA-Moleküle so geführt, dass sie schließlich zu einem zehnten Protein gelangen, welches als katalytische Untereinheit die RNA in Stücke schneidet“, beschreibt Debora Makino die Funktionsweise. Das Fass ist essentiell für den Abbau-Prozess, weil es dazu beiträgt, dass die RNA entfaltet und für die Zerkleinerung vorbereitet wird. „Zellen, denen eines dieser zehn Proteine fehlt, sind nicht überlebensfähig. Das zeigt, dass nicht nur die katalytische Untereinheit, sondern auch das gesamte Fass für die Funktion des Exosoms essentiell sind“, erklärt Makino.
Das Anbinden von RNA und Führen der RNA durch den Kanal des Exosoms geschieht in Eukaryoten in ähnlicher Weise wie in Bakterien und Archaebakterien, welche die Wissenschaftler bereits in früheren Arbeiten strukturell untersucht haben. „Obwohl der eigentliche Abbau chemisch sehr unterschiedlich in Eukaryoten und Bakterien beziehungsweise Archaebakterien abläuft, wird die RNA auf gleiche Weise durch den Kanal befördert. Vergleichbar ist der Mechanismus auch mit dem des Proteasoms, einem Komplex für den Protein-Abbau“, sagt Elena Conti. In Zukunft wollen die Wissenschaftler verstehen, wie das Exosom gezielt zu den RNA-Molekülen gelangt, welche für den Abbau vorgesehen sind, und wie es in den unterschiedlichen Bereichen der Zelle reguliert wird.
Originalpublikation
Makino, D.L., Baumgärtner, M., Conti, E.: Crystal Structure of an 11-Subunit Eukaryotic Exosome Complex Bound to RNA. Nature, February 3, 2013.
DOI: 10.1038/nature11870
Kontakt
Prof. Dr. Elena Conti
Zelluläre Strukturbiologie
Max-Planck-Institut für Biochemie
Am Klopferspitz 18
82152 Martinsried
Email: conti[a]biochem.mpg.de
Anja Konschak
Öffentlichkeitsarbeit
Max-Planck-Institut für Biochemie
Am Klopferspitz 18
82152 Martinsried
Tel.: +49 (0) 89 8578-2824
Email: konschak[a]biochem.mpg.de
www.biochem.mpg.de












