Was Chromosomen im Innersten zusammenhält
Max-Planck-Forscher klären Funktionsweise von DNA-Verpackungsproteinen auf
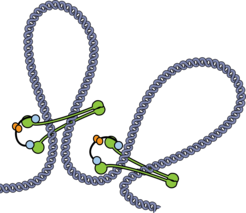
Der SMC-Proteinkomplex (grün, blau, orange) umfasst die DNA (grau) wie eine Klammer und sorgt so für Ordnung.
In jeder menschlichen Zelle müssen etwa zwei Meter DNA in einem Zellkern untergebracht werden, der nur einen Durchmesser von wenigen Tausendstel Millimetern hat. Die DNA ist im Zellkern in einzelnen Chromosomen organisiert. Um für einen sicheren Transport der DNA bei der Zellteilung zu sorgen, müssen diese langen und verknäulten DNA-Fäden dicht verpackt werden.
Dieser Schritt ist bisher nur in groben Ansätzen verstanden. Eine Schlüsselrolle spielen hierbei die SMC-Proteinkomplexe, die aus zwei Proteinarmen (SMC) und einem Bindeglied (Kleisin) bestehen. Gemeinsam bilden sie eine ringartige Struktur. „Wie wichtig diese Proteinkomplexe sind, wird auch dadurch deutlich, dass der SMC-Komplex vom Bakterium bis zum Menschen ähnlich aufgebaut ist und funktioniert“, erläutert Larissa Wilhelm, Doktorandin am MPI für Biochemie.
Es gibt verschiedene Möglichkeiten, wie der SMC-Komplex DNA verpacken kann. Er könnte die einzelnen DNA-Fäden zum Beispiel zusammenkleben. Wie die Max-Planck-Forscher jedoch in Bakterien zeigen konnten, legen sich die SMC-Proteinarme wie eine Klammer um die DNA und können so zum Beispiel weit entfernte Teile der DNA zusammenbringen und das Chromosom zu einer kompakteren Form führen.
In einem nächsten Schritt wollen die Wissenschaftler der Forschungsgruppe „Chromosomale Organisation und Dynamik“ jetzt untersuchen, ob sich die Klammer kurz öffnet und bereits in Schlaufen gelegte DNA umklammert oder ob sie erst an die DNA bindet und dann selbst DNA-Schlaufen bildet, indem sich die Klammer über die DNA stülpt. „Unsere Ergebnisse könnten auch helfen, die komplexe Organisation von menschlichen Chromosomen zu verstehen und damit Einblicke in die Entstehung von Erbdefekten wie Trisomie 21 gewähren“, sagt Stephan Gruber, Gruppenleiter am MPI für Biochemie.
Originalpublikation
L. Wilhelm, F. Bürmann, A. Minnen, H.-C. Shin, C.P. Toseland, B.-H. Oh, S. Gruber: SMC condensin entraps chromosomal DNA by an ATP hydrolysis dependent loading mechanism in Bacillus subtilis. eLIFE, May 7, 2015.
DOI: 10.7554/eLife.06659

