Krebs verstehen
Forschungsbericht (importiert) 2022 - Max-Planck-Institut für Biochemie
Molekulare Arbeitspferde
Um nachzuvollziehen, wie wir zu dem Entschluss kamen, vier verschiedene Methoden in einer einzigen Technologie zusammenzufassen, muss man zunächst verstehen, dass Proteine die wichtigsten Puzzlestücke für eine Vielzahl von Erkrankungen sind. Proteine werden auch als die „molekularen Arbeitspferde“ der Zelle bezeichnet. Ihre korrekte Funktion entscheidet über die Funktionsfähigkeit einer Zelle und damit auch über das Funktionieren eines ganzen Individuums. Wenn also etwas in unseren Zellen nicht richtig funktioniert und wir krank werden, kann man sicher sein, dass insbesondere unsere Proteine daran beteiligt sind. Aus diesem Grund kann die Kartierung der Proteinlandschaft, also ihrer individuellen Zusammensetzung, dabei helfen herauszufinden, warum sich beispielsweise ein Tumor in einem Patienten entwickeln konnte und welche Schwachpunkte dieser Tumor hat. Darüber hinaus können wir durch diese Erkenntnisse weiter schlussfolgern, welche Behandlungsmethoden bei bestimmten Patientinnen und Patienten von Vorteil sein könnten.
Angeregt durch diese Fragen und neuen Möglichkeiten für Krebstherapien haben wir eine neue, innovative Methode entwickelt. Deren Bezeichnung, Deep Visual Proteomics, leitet sich aus dem Englischen von unserer Vorgehensweise ab: Wir verwenden Deep-Machine-Learning-Algorithmen, um Zellen in visuell hochauflösenden Bildern zu erkennen, diese mittels eines Lasermikroskops zu extrahieren und mit Methoden der Proteomik zu kombinieren. Dies erlaubt uns, die Vielzahl und Zusammenarbeit verschiedener Zellen genauer zu erforschen und vieles Neues über die Entstehung von Krebs zu lernen.
Unsere Technologie stellt Forschenden sowie Klinikerinnen und Klinikern somit Informationen zur Verfügung, die auf der Zusammensetzung von Proteinen beruhen. Mit diesen Erkenntnissen können Onkologinnen und Onkologen gezielte Strategien für Diagnosen und für neue oder gar personalisierte Therapien erstellen.
Die vier Methoden hinter Deep Visual Proteomics
Mit Deep Visual Proteomics haben wir die Vorteile von i.) moderner Mikroskopie, ii.) maschinellem Lernen, iii.) Lasermikrodissektion und iv.) ultra-sensitiver Massenspektrometrie mit nachfolgenden bioinformatischen Analysen kombiniert. Mithilfe der modernen Mikroskopie werden zunächst hochauflösende Gewebekarten erstellt, man kann sich das wie eine Art Landkarte vorstellen. Mit diesen Landkarten werden durch maschinelles Lernen und künstliche Intelligenz die in der Karte vorkommenden Zellen in verschiedene Klassen eingeordnet. Für diese Einordnung werden die Zellen hinsichtlich ihrer Form, Größe oder Proteinlokalisierung betrachtet. Im Anschluss daran werden die Zellen durch hoch akkurate Lasermikrodissektion aus dem Gewebe herausgetrennt und in gesunden oder verschiedenen erkrankten Zellgruppen sortiert gesammelt. In den einzelnen Zellgruppen werden dann Tausende von Proteinen gleichzeitig mittels ultra-sensitiver Massenspektrometrie erkannt und analysiert, um zuletzt aus diesen Ergebnissen mittels bioinformatischer Analysen Proteinkarten zu erstellen (Abb. 1; [1]).
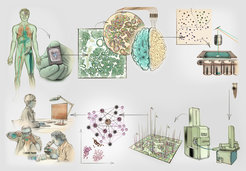
Auf den Proteinkarten wird die Verteilung der Proteine in komplexen Krankheiten wie Krebs wiedergegeben. Sie stellen ein neues, wertvolles Hilfsmittel für Klinikerinnen und Kliniker dar, mit dem die Mechanismen von Gesundheit und Krankheit besser verstanden werden können. Durch unser neues Konzept könnte somit auch ein Wandel in der wissenschaftlichen Grundauffassung der molekularen Pathologie in der Klinik entstehen. In der Studie, in der wir unsere Technologie erstmals angewendet haben, konnten visuelle Merkmale eines Tumors bestimmt und Proteine in denjenigen Krebszellen untersucht werden, die direkt an gesundes Gewebe angrenzten. Man könnte also beispielsweise eine Gewebeprobe aus einem Tumor entnehmen, innerhalb kürzester Zeit und mit geringem Aufwand die Gesamtheit der Proteine bestimmen und die Mechanismen erkennen, die am Voranschreiten der Tumorentwicklung beteiligt sind. Durch eine einzige Gewebeanalyse eines Patienten könnten so schnell und gezielt therapeutische Ansätze abgeleitet werden.
Medizinischer Fortschritt dank Deep Visual Proteomics
Im Verlauf unserer Studie konnten wir Deep Visual Proteomics auf Zellen von Speicheldrüsen- und Hautkrebs Patientinnen und Patienten anwenden. Dabei gelang es, einen klinisch hochkomplexen Fall zu diagnostizieren. Die bisher mit unserer Methode erzielten Erkenntnisse und die neuen Chancen, die sich dadurch eröffnen, lassen uns hoffen, dass künftig Therapieformen gegen Tumore entwickelt werden können, für die es bisher noch keine gibt.
Unsere Technik begrenzt sich nicht nur auf die von uns bereits untersuchten Krebsarten, sondern ist prinzipiell auf eine Vielzahl von Tumoren und anderen Erkrankungen anwendbar. So könnten mit Deep Visual Proteomics beispielsweise auch neurodegenerative Erkrankungen wie Alzheimer oder Parkinson untersucht werden.
Durch die Kombination von Mikroskopie, künstlicher Intelligenz und hochempfindlicher Massenspektrometrie basierter Proteomik haben wir eine leistungsfähige Technologie entwickelt. Sie wird uns dabei helfen, die molekulare Verschaltung gesunder und kranker Zellen verstehen zu lernen. Wir hoffen, dass Deep Visual Proteomics auch Ärztinnen und Ärzten dabei helfen wird, neue Angriffspunkte für zukünftige Medikamente und Diagnosen zu identifizieren. Unsere Technologie hat das Potenzial, den medizinischen Fortschritt weiter voranzutreiben.
