Steuerung der Proteinproduktion durch kleine RNA-Moleküle
Forschungsbericht (importiert) 2009 - Max-Planck-Institut für Biochemie
Kleine nichtkodierende RNAs – grundlegende Regulatoren der Genexpression
Nach der Entschlüsselung des humanen Genoms wurde schnell klar, dass nur ein sehr kleiner Teil der menschlichen genetischen Information Proteine kodiert. Der weitaus größere Teil des humanen Genoms (man schätzt mehr als 90%) ist nichtkodierender Natur und man ging zunächst davon aus, dass es sich dabei um „Müll-DNA“ handelt. Inzwischen ist aber bekannt, dass fast die gesamte nichtkodierende DNA ständig in nichtkodierende RNA umgeschrieben (transkribiert) wird und die so entstandenen RNA-Moleküle für die korrekte Funktion von Zellen von entscheidender Bedeutung sind. Aufgrund intensiver Forschung in den letzten Jahren wissen wir heute, dass nichtkodierende RNAs auch an der Entstehung und Ausprägung von verschiedenen Krankheiten wie neurodegenerative Erkrankungen, aber auch Krebs, beteiligt sind. Eine sehr wichtige Klasse dieser RNAs sind so genannte kleine nichtkodierende RNAs, die durch ihre charakteristische Länge von 20-30 Nukleotiden gekennzeichnet ist. Wichtige Vertreter solcher RNAs sind zum Beispiel microRNAs (miRNAs) oder small interfering RNAs (siRNAs). Diese RNAs regulieren die Übersetzung von DNA in Proteine an unterschiedlichen Stellen und sind somit wichtige Regulatoren in der Zelle; siRNAs waren die ersten regulatorischen RNAs, die entdeckt wurden, und sie sind für das inzwischen bekannte Phänomen der RNA-Interferenz (RNAi) verantwortlich.
RNA-Interferenz (RNAi)
RNAi wurde zunächst im Fadenwurm Caenorhabditis elegans und in Pflanzen entdeckt und beschreibt das Phänomen, dass doppelsträngige RNA (dsRNA) komplementäre RNA-Moleküle erkennen und deren Abbau induzieren kann [1]. Genaue Untersuchungen haben gezeigt, dass das zelluläre Enzym Dicer lange doppelsträngige RNA in kurze, etwa 21 Nukleotide lange doppelsträngige RNAs spaltet, die siRNAs genannt wurden. SiRNAs sind durch 5’-Phosphate und zwei Nukleotide lange 3’-Überhänge charakterisiert. Die doppelsträngige siRNA wird in der Zelle entwunden und ein Strang (guide strand) wird in den RNA-induced silencing complex (RISC) eingebaut. Der andere Strang (oft als passenger strand bezeichnet) wird mittels zellulärer Abbausysteme entsorgt. Im Wesentlichen wird von der thermodynamischen Stabilität der RNA-Enden bestimmt, welcher der beiden siRNA-Stränge in RISC eingebaut wird. Der Strang, dessen 5’-Ende weniger stark gepaart ist, wird in der Regel präferenziell in funktionelle RISC-Komplexe inkorporiert. Mithilfe der eingebauten siRNA erkennt RISC vollständig komplementäre einzelsträngige RNAs und spaltet diese [2].
Neben der mittlerweile zahlreichen Anwendungsmöglichkeiten in der biologischen Grundlagenforschung spielt RNAi auch eine sehr wichtige Rolle bei der Entwicklung von Medikamenten. Bei vielen Krankheiten sind so genannte Krankheitsgene zu stark exprimiert und es wird versucht, mittels RNAi die Expression solcher Gene zu unterdrücken, ohne dass man dabei direkt in das Genom via Genmanipulation eingreift. RNAi-basierte Medikamente befinden sich bereits in fortgeschrittenen klinischen Phasen und werden sicherlich in den nächsten Jahren breite Anwendung finden [3].
Die Argonaute-Protein-Familie
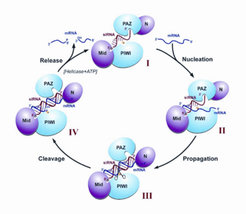
Mitglieder der Argonaute-Proteinfamilie stellen die wichtigsten Proteinkomponenten von RISC dar. Beim Menschen existieren acht Ago-Proteine, wobei nur Ago1-4 ubiquitär, das heißt in allen menschlichen Zellen, exprimiert werden. Argonaute-Proteine sind durch eine PAZ-, PIWI- sowie eine MID-Domäne gekennzeichnet. Während die MID-Domäne das 5’-Ende der kleinen RNA bindet, ist das 3’-Ende in der PAZ-Domäne verankert. Die PIWI-Domäne zeigt strukturelle Ähnlichkeit zur RNase H, und biochemische Untersuchungen haben gezeigt, dass humanes Ago2 die Endonuklease (ein RNA-spaltendes Enzym) von RISC darstellt, welche die komplementäre RNA sequenzspezifisch spaltet. Dieses Enzym wurde, in Anspielung auf Dicer, daher „Slicer“ genannt (Abb. 1). Interessanterweise besitzen Ago1, Ago3 und Ago4 keine Slicer-Aktivität, obwohl sie Ago2 sehr ähnlich sind [4].
MicroRNAs und die Feinregulation der Genexpression
Während siRNAs in der Regel aus von außen in die Zelle eingeschleuster RNA gebildet werden, werden miRNAs von spezifischen Genen endogen produziert. Dabei werden miRNA-Gene zunächst als primäre miRNA-Transkripte durch die RNA-Polymerase II im Zellkern transkribiert. Der microprocessor-Proteinkomplex, der als katalytische Komponente das RNase-III-Enzym Drosha enthält, spaltet die primären miRNA-Transkripte und generiert miRNA-Vorläufermoleküle (pre-miRNAs). Pre-miRNAs sind durch eine ausgeprägte hairpin-Struktur gekennzeichnet und werden ins Cytoplasma transportiert, wo das RNase-III-Enzym Dicer die pre-miRNAs in 20–23 Nukleotide lange doppelsträngige RNAs (dsRNAs) spaltet. Die kurzlebigen dsRNAs werden schließlich entwunden, und einzelsträngige, reife miRNA wird in spezifische Protein-Komplexe, sogenannte miRNPs, eingebaut (Abb. 2) [5].
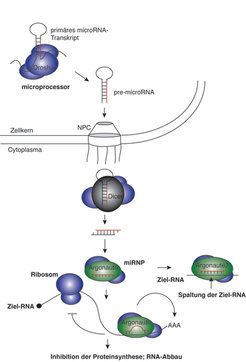
Auch miRNAs können wie siRNAs wirken und die Spaltung von vollständig komplementären RNAs steuern. Während pflanzliche miRNAs hauptsächlich die Spaltung von Ziel-RNAs bewirken, wurden nur wenige tierische miRNAs mit vollständiger Komplementarität zu zellulären RNAs entdeckt. Tierische miRNAs interagieren hauptsächlich mit unvollständig komplementären Bereichen in den 3’-untranslatierten Regionen (UTR) von mRNAs und inhibieren deren Translation. Darüber hinaus wurde kürzlich ein weiterer Mechanismus der miRNA-vermittelten Genregulation beschrieben. Hierbei rekrutieren miRNAs spezifische Deadenylasen, die das Poly-A-Ende der mRNA verkürzen. Dies führt schließlich zum Abbau der mRNA und folglich zur Inhibition der Genexpression (Abb. 2) [6].
Computergestützte Analysen haben gezeigt, dass einzelne miRNAs eine Vielzahl von Ziel-RNAs regulieren können. Zahlreiche Studien haben weiterhin herausgefunden, dass miRNAs die Expression ihrer Zielgene nicht vollständig blockieren. Vielmehr unterdrücken viele miRNAs die Proteinproduktion nur um maximal 60-70%. Man geht daher davon aus, dass viele miRNAs die Expression ihrer Zielgene lediglich feinregulieren und somit die richtige Menge der entsprechenden Proteine für die Zelle garantieren.
MiRNAs stellen wichtige zelluläre Schaltstellen dar und es ist daher nicht verwunderlich, dass sie auch wichtige Rollen bei der Entstehung von Krankheiten, wie zum Beispiel Krebs, spielen. Je nach Ziel-Gen, welches von der miRNA reguliert wird, kann eine miRNA dabei als Onkogen oder als Tumorsuppressor wirken. Die spezifische Inhibition solcher miRNAs als Therapieform ist daher Gegenstand intensivster Forschung.
Identifizierung von neuartigen miRNA-Genen
Durch die Verfügbarkeit immer besserer und sensitiverer Sequenziermethoden ist es gelungen, weitere Klassen von kleinen RNAs zu finden. So konnte gezeigt werden, dass eine Klasse von nichtkodierenden RNAs, so genannte small nucleolar RNAs (snoRNAs), zu kleinen RNAs prozessiert werden kann, die dann wiederum wie miRNAs wirken können [7]. SnoRNAs sind im Zellkern zu finden, wo sie innerhalb der Nukleoli angereichert vorliegen. Nukeoli sind an der Biogenese der Ribosomen beteiligt, bei der snoRNAs eine wichtige Rolle spielen. Ein kleiner Teil dieser snoRNAs kann aber auch ins Cytoplasma gelangen und dort von Dicer zu kleinen RNAs weiterverarbeitet werden (Abb. 3).
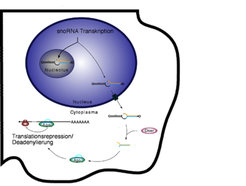
Inzwischen gibt es Hinweise, dass snoRNAs bei der Entstehung einer neurodegenerativen Krankheit, dem sogenannten Prader-Willi-Syndrom, eine wichtige Rolle spielen könnten. Es ist denkbar, dass nicht die snoRNAs selbst, sondern die daraus entstehenden kleinen RNAs dabei entscheidend sind. Sehr wahrscheinlich ist, dass es neben den beschriebenen noch eine Vielzahl bislang unentdeckter Klassen nichtkodierender RNAs gibt. Zukünftige Arbeiten auf diesem Gebiet dürften nicht nur zu einem besseren Verständnis der menschlichen Zelle beitragen, sondern auch zu neuen Erkenntnissen über die molekularen Ursachen verschiedener Krankheiten führen.


