Wie die Zelle dem Chaos Zügel anlegt
Forschungsbericht (importiert) 2010 - Max-Planck-Institut für Biochemie
Der Blick in die lebende Zelle
Die Mikroskopie stellt seit mehreren Jahrhunderten eine Schlüsseltechnologie der biologischen Forschung dar. Einen Grundstein legte im 17. Jahrhundert der niederländische Mikroskopbauer Antoni van Leeuwenhoek, dessen Geräte bereits Bakterien sichtbar machen konnten. Lichtmikroskope der heutigen Generation enthalten perfektionierte Linsen, die Vergrößerungen nahe den physikalisch gesetzten Grenzen ermöglichen. Sie erlauben die Abbildung kleinster zellulärer Strukturen und sind für die Forschung im Bereich der Zellbiologie unabdingbar. Ein bahnbrechender Schritt für die Mikroskopie an lebenden Zellen – im Jahr 2008 mit dem Nobelpreis für Chemie gewürdigt – war die Entdeckung des grün fluoreszierenden Proteins GFP [1]. Dieses Molekül stammt aus der Qualle Aequorea victoria und hat zwei für die Forschung unschätzbare Eigenschaften: Zum einen kann es mit einer Vielzahl zellulärer Proteine gekoppelt werden. Hinzu kommt, dass GFP unter Anregung mit blauem oder ultraviolettem Licht fluoresziert. Auf diese Weise verrät es den Forschern auch den Aufenhaltssort desjenigen Moleküls, mit dem es fusioniert ist.
GFP war nur der erste in einer Reihe vielseitig einsatzbarer biologischer Marker, die unter anderem analysieren helfen, wo und in welcher Menge sich ein Protein zu einem bestimmten Zeitpunkt in der Zelle befindet. Für die Forschung war dieser Nachweis zuvor oft wie die Suche nach einer Nadel im Heuhaufen, präsentiert sich die Zelle doch auf den ersten Blick als koordiniertes Chaos aus einer Unmenge molekularer Strukturen. Um zu analysieren, was die Aufnahmen überhaupt zeigen, müssen als Dritter im Bunde neben der modernen Mikroskopie- und Kameratechnik auch bildverarbeitende Verfahren zum Zuge kommen. Sie weisen mit fast mathematischer Präzision nach, wie biologische Prozesse in der Zelle ablaufen. Mit bloßem Auge ist nämlich oft nicht erkennbar, was sich aber zwingend aus komplexen Berechnungen ergibt: Vom Chaos findet sich in der Zelle keine Spur; sie ist vielmehr straff und effizient organisiert.
Das wird immer dann deutlich, wenn mithilfe der neuen Methoden bislang unbekannte zelluläre Strukturen nachgewiesen werden können. Ein Beispiel ist die Plasmamembran, die als Barriere und Kontaktstelle zur Außenwelt der Zelle gleichermaßen wichtige Funktionen erfüllt. Sie besteht aus einer Vielzahl von Lipiden und Proteinen, die auf dem flüssigen Zellinneren „schwimmen“ und dabei – so wurde bislang jedenfalls vermutet – über ein hohes Maß an Bewegungsfreiheit verfügt. Erst jetzt konnte aber gezeigt werden, dass die Membran von Hefezellen nicht gleichförmig, sondern strukturiert ist – wobei unterschiedliche Proteine in dynamischen, netzwerkartigen Bereichen residieren (Abb. 1). Ermöglicht wurde diese Einsicht durch den Einsatz ausgefeilter optischer Methoden wie etwa TIRFM, kurz für total internal reflection microscopy, die hoch aufgelöste Aufnahmen an und direkt unter der Zellmembran erlaubt. Eine weitere Technik, die fluorescence recovery after photobleaching, oder oder kurz FRAP, erlaubt, die Bewegung von Molekülen zu messen und quantitativ zu erfassen.
Ein dynamisches Gerüst im Rampenlicht
Erst jetzt, mit dem geeigneten Instrumentarium an der Hand, zeichnet sich ab, wie wichtig eine Erfassung der Bewegung von Zellbausteinen für das Verständnis zellulärer Prozesse ist. Einen entscheidenden Anteil an dieser Dynamik trägt dabei ein zentraler Bestandteil aller höheren Zellen: das Zytoskelett. Es verleiht der Zelle unter anderem ihre Form, stützt sie und wird zudem als mikroskopisch kleines Schienensystem für den Transport molekularer „Güter“ bei nahezu allen zellulären Prozessen herangezogen. Dabei sorgt das Zytoskelett nicht nur für Bewegung, sondern ist als flexibles Geflecht aus Filamenten auch selbst hochdynamisch.
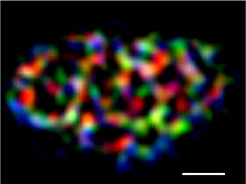
Eine Hauptkomponente des Zytoskelets ist Aktin; viele kugelförmige Aktinmonomere lagern sich zu langen Filamenten zusammen. Bis vor kurzem war die Markierung von Aktinfilamenten in lebenden Zellen nur mit Substanzen oder Sonden möglich, die entweder kompliziert in der Handhabung waren oder die Dynamik der Aktinmoleküle beeinträchtigten. Es war deshalb ein großer Fortschritt, als hier die Entwicklung eines fast universell verwendbaren Markers gelang, der die zellulären Prozesse nicht verfälscht [2]. Dieser Marker, lifeact genannt, wird mittlerweile in Kombination mit verschiedenen Fluoreszenfarbstoffen zur Untersuchung des Aktinzytoskeletts in einer Vielzahl von pflanzlichen und tierischen Zellen eingesetzt (Abb. 2). Der Grundlagenforschung wie auch der Biomedizin stehen damit ganz neue Optionen offen, um das Aktin-Protein und all seine Funktionen zu analysieren – von denen es eine ganze Reihe gibt. So wirken Aktinfilamente bei der Teilung, Bewegung, mechanischen Stabilisierung und Formgebung von Zellen mit. Aktin spielt aber auch eine zentrale Rolle bei vielen Krankheiten, wie Muskeldystrophien oder Neurodegenerativen Erkrankungen [3]. Ohne Aktin aber würden Zellen zum Stillstand kommen und in sich zusammenfallen.
Gut vernetzt

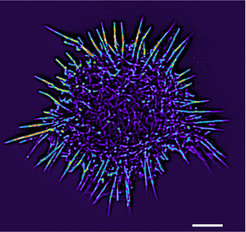
Meist treten Aktinfilamente in größeren Verbänden auf. Sie können sogar Netzwerke bilden. Einzelne Filamente in diesem Geflecht werden ständig neu gebildet und ebenso schnell wieder abgebaut. Manchmal durchlaufen sie dabei eine Art Zyklus. Dann werden an ihrem Vorderende neue Aktinmoleküle angefügt, während am Hinterende entsprechend Untereinheiten wegfallen. Das Filament scheint sich dann – gleichzeitig wachsend und schrumpfend – durch die Zelle zu bewegen. Ein Netzwerk aus solchen „Aktintretmühlen“ entwickelt ausreichend Kraft, um die Zellmembran vorwärts schieben zu können und ermöglichen Zellen dadurch eine fließende Bewegung, also Mobilität – wie sie etwa für weiße Blutkörperchen bei der Immunabwehr charakteristisch ist [4]. Dabei ist noch lange nicht restlos geklärt, welche Aktinstrukturen sich in Zellen bilden können: Neue Beobachtungen an der Oberseite von Epithelzellen aus Säugetieren offenbarten wurmähnliche Aktinbündel (Abb. 3) mit ungewöhnlicher Dynamik: Diese Strukturen sind in ständiger Bewegung und zeigen eine hohe Plastizität mit häufigen Fusionen oder Spaltung von Bündeln in mehrere kürzere Stücke. Gleichzeitige Markierung von Aktin und dem in der Muskelkontraktion wichtigen Myosin zeigen, dass die Zelloberfläche komplett von einem gemeinsamen Netzwerk dieser beiden Proteine bedeckt ist. Vermutlich dient dieses Geflecht dazu, den Einfluss externer Kräfte auf die Zelle auszugleichen. Diese wirken vor allem während der Zellwanderung oder bei größeren Umlagerungen von Geweben und Zellverbänden, etwa während der Embryonalentwicklung oder bei der Verbreitung von Krebszellen im Körper. In welchem Maße sich mechanische Kräfte und andere Faktoren der Umgebung auf biologische Systeme auswirken, ist mittlerweile sogar Gegenstand eines eigenen Forschungszweiges – der Mechanobiologie. So weiß man, dass sich noch undifferenzierte Stammzellen auf einem harten Untergrund eher zu Knochenzellen, auf einer etwas weicheren Unterlage zu Muskelzellen und in ganz weicher Umgebung zu Nervenzellen entwickeln [5].
Das Skelett der Bakterien
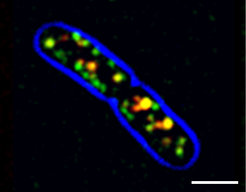
Für seine erstmalige Beschreibung von Bakterien im menschlichen Speichel erntete Antoni van Leeuwenhoek noch großen Spott. Heute aber kennt man nicht nur eine Vielfalt an Bakterien, sondern kann sogar beobachten, was sich im Detail in ihnen abspielt. Dabei zeigte sich erst vor wenigen Jahren, dass das Zytoskelett wohl sehr früh in der Evolution entstanden ist, weil sich in Bakterien eine Entsprechung findet. MreB etwa heißt eine aktinähnliche Komponente, die bei der Organisation der bakteriellen Zellwand eine wichtige Rolle spielt. Bislang wurde vermutet, dass sich MreB-Filamente zu einer langen Wendel zusammenlagern, die die Bakterienzelle durchzieht und dadurch Proteine für die Zellwandsynthese positioniert [6]. Untersuchungen am Bakterium Bacillus subtilis – auch hier mithilfe von TIRFM und FRAP – zeigten jetzt aber, dass MreB-Filamente lediglich kleine Plattformen bilden, die sich auf Kreisbahnen entlang der Zellmembran bewegen (Abb. 4). Überraschenderweise wird diese Bewegung allerdings nicht durch Motorproteine oder die Dynamik des Zytoskeletts angetrieben, sondern direkt von der Zellwandsynthese. Dieser Fund widerlegt nicht nur lang gehegte Modelle zum Aufbau der Zellwand, sondern könnte durchaus auch von praktischem Nutzen sein: Viele Antiobiotika wirken auf die bakterielle Zellwand. Ein besseres Verständnis der hier zugrunde liegenden Vorgänge könnte zur Entwicklung neuer Wirkstoffe und zur Vermeidung zunehmender Resistenzen bei Erregern beitragen.
Ausblick
Vom Bakterium bis zur Säugerzelle: In vielen interdisziplinär arbeitenden zellbiologischen Arbeitsgruppen werden vielfältige Ansätze aus der Biologie, Physik, Chemie, Mathematik und Informatik kombiniert. Nur so lassen sich die komplexen Grundlagen vieler biologischer Prozesse mit ausreichender Präzision analysieren und auch quantitativ erfassen. Modelle liefern dabei die theoretische Grundlage und Ergänzung zur mikroskopischen Untersuchung an den diversen biologischen Objekten. Transportiert, gebaut, umgebaut und abgebaut wird in allen Zellen. Die molekularen Großbaustellen mit ihrer verborgenen Organisation finden sich nicht zuletzt auch in uns.



