Minimalisierung von Lebensprozessen
Forschungsbericht (importiert) 2015 - Max-Planck-Institut für Biochemie
Synthetische Biologie in biophysikalischer Perspektive
Unsere Kenntnis dessen, welche Prozesse in lebenden Zellen ablaufen und auf welchen Molekülen sie beruhen, hat in den letzten 50 Jahren enorme Ausmaße angenommen. Wir verstehen immer besser, was in der Informationszentrale der verschiedenen Zellen, ihrer DNA, geschrieben steht, welche komplexen Proteinmaschinen in ihnen hergestellt werden und haben einen Begriff von der Breite und Vielfalt der Interaktionsnetzwerke dieser Proteine. Bereits in einfachen Zellen ähneln diese Netzwerke mit ihrer Vielzahl von Akteuren, Wechselwirkungen und Schaltstellen, an denen verschiedene Prozessketten zusammenlaufen, durchaus dem Internet als dem vermutlich komplexesten Netzwerk, das wir heute kennen.
Unsere modernen Analysemethoden, die uns mittlerweile bereits erlauben, riesige Proteine bis auf ihre Einzelatome herab abzubilden oder einzelnen Molekülen in der Zelle bei der Arbeit zuzuschauen, legen es nahe, über die Biologie immer mehr auch in technologischen Konzepten nachzudenken, sie also wie jedes technische System in einzelne wohldefinierbare und verstehbare Funktionsmodule zu zerlegen. Mit dem zunehmenden Wissen über diese Module und deren Handhabung entsteht wie in anderen wissenschaftlichen Disziplinen zuvor - das letzte Mal in der Chemie vor etwa 100 Jahren - der Drang, nicht nur zu analysieren, sondern diese Module ganz neu zu kombinieren, also nicht nur Analyse, sondern auch Synthese zu betreiben. Wie damals aus der analytischen die synthetische Chemie entstanden ist, der wir einen nicht unerheblichen Teil der uns umgebenden Welt verdanken, so entsteht gegenwärtig aus der analytischen Biologie die synthetische Biologie.
Diese Synthetische Biologie kann man nun mit verschiedenen Zielen betreiben. Das eine Ziel, das insbesondere aus der angewandten Biotechnologie kommt, ist es, lebende Organismen, die als Produktionssysteme für neue Medikamente, Chemikalien, Materialien oder Energie eingesetzt werden, so zu verändern, dass deren Herstellung effizienter oder überhaupt erst möglich wird. Das andere Ziel ist fundamentaler, es beruht auf der Frage, ob es so etwas wie einen Basissatz von Funktionsmodulen, also Molekülen und ihren Wechselwirkungen, gibt, mit dem Leben überhaupt beginnt. Diese Spielart nennen wir bottom-up, also Synthese lebender Systeme von unten, aufbauend auf einzelnen molekularen Bausteinen (Abb. 1). Ob so etwas im Labor überhaupt möglich ist, ist ungewiss, allerdings hat die Natur selbst diesen Schritt vor etwa 3,5 Milliarden Jahren vollzogen.
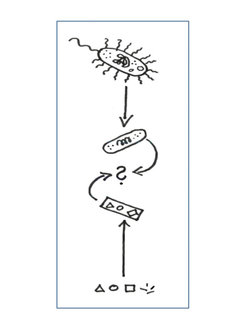
Die Attraktivität des bottom-up Ansatzes für die Biophysik ist offensichtlich: Erstmals wären nicht nur Teilsysteme und Aspekte lebender Zellen quantitativ und durchgängig erfassbar, sondern auch das Gesamtsystem der - minimalen - Zelle. Erst dann könnte man von einem wirklichen physikalischen Verständnis des Phänomens Leben reden. Bis dahin ist es aber noch ein weiter Weg. Nicht nur kennen wir die Moleküle und Bedingungen nicht mehr, die auf der Erde oder sonst im Universum an der Entstehung des Lebens beteiligt waren, sondern unsere heutigen Organismen haben sich im Laufe dieser langen Zeit durch Evolution auch so weit davon fortentwickelt, dass eine rückwärtige Extrapolation praktisch unmöglich ist. Der einzige Ansatzpunkt ist der Versuch, bekannte biologische Teilsysteme und Phänomene ihrerseits auf eine nicht Organismus spezifische Quintessenz hin zu analysieren. Dies geschieht durch eine sogenannte Rekonstitution, also den Transfer eines Prozesses in zellfreie Systeme, wo er isoliert vom zellulären Kontext analysiert und verstanden werden kann.
Ein minimaler Zellteilungsapparat?
Welche Funktionen lebender Systeme mit Hilfe eines bottom-up Ansatzes zuerst nachgestellt werden sollen, hängt natürlich von der wissenschaftlichen Schwerpunktsetzung ab. Unsere Forschung hat sich zunächst eine Rekonstitution der Zellteilung zum Ziel gesetzt. Welche Minimalausstattung von (Protein-) Funktionselementen ist hierfür nötig? Die Hülle der meisten heutigen Zellen besteht im Wesentlichen aus einer Membran - einer Sandwichstruktur aus so genannten Lipiden. Lipide sind Fettsäuremoleküle mit einem wasserlöslichen und einem wasserunlöslichen Ende. Lagern sie sich in wässriger Umgebung spontan zusammen, entstehen seifenblasenähnliche Vesikel. In diesen Blasen können sich alle Stoffe sammeln, die auch in Wasser löslich sind. Die Membranblasen sind zwar sehr weich und leicht verformbar, aber praktisch undurchdringbar für größere Moleküle. Die ideale Struktur also, um eine minimale Zelle zu begrenzen. Doch wie können sich jetzt in diesen einfachen Seifenblasen so etwas wie Strukturen ausbilden? Wie können die eingeschlossenen Moleküle erfassen, was und wie groß genau ihre Hülle ist und vor allem: Wie können sie diese Hülle zur Teilung bringen?
Diese Aufgaben sind in allen heute bekannten Zellen auf so komplizierte Weise geregelt, dass der Nachbau einer dieser Zellteilungsmaschinerien, auch schon von primitiven Mikroorganismen, eine Herkulesaufgabe zu sein scheint. Und doch gibt es Motive und Moleküle, die so interessant und vielversprechend sind, dass sie sich als Modelle zum Nachbau geradezu anbieten. So zeigt zum Beispiel das Bakterium Escherichia coli, eines der „Haustierchen“ der modernen Biotechnologie, eine höchst bemerkenswerte Abfolge von Prozessen bei der Zellteilung: Das Bakterium ist stäbchenförmig und wächst in Längsrichtung, bis es ab einer bestimmten Länge einen sogenannten Teilungsring aus Proteinen ausbildet, der sich durch eine bislang nicht verstandene Kraft zusammenzieht und die Zelle in zwei Tochterzellen abschnürt. Wie „weiß“ nun aber dieser Ring, wo die Mitte ist, und wie kommt er dort hin?
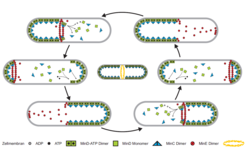
Der Positionierung dieses bakteriellen Teilungsrings liegt ein spektakulärer Musterbildungsmechanismus zugrunde. Hierfür organisieren sich die sogenannten MinDE Proteine unter Nutzung der zellulären Energiewährung ATP derart, dass sie im Minutentakt zwischen den beiden Polen des Bakteriums hin und her oszillieren. Durch diese Oszillation entsteht im zeitlichen Mittel eine höhere Konzentration an den Polen als in der Mitte. Und ein drittes Protein MinC sorgt dafür, dass sich nur dort, wo die anderen Proteine nicht sind, der Ring anlagern kann, also in der Mitte. Durch die unterschiedliche „Laufzeit“ der Oszillationen kann die Zelle auch genau bestimmen, bei welcher Länge sie eine Teilung durchführt (Abb. 2).
Dass der Ausbildung dieser Oszillationen ein sehr einfacher Reaktionsmechanismus zugrunde liegt, der tatsächlich nur auf der Wechselwirkung zweier verschiedener Molekülarten, einer Membran und der Energiewährung ATP beruht, konnten wir in den letzten Jahren durch Rekonstitution von MinD und MinE auf künstlichen Membranen zeigen. Auf nicht begrenzten Oberflächen entstehen anstelle von Oszillationen fortschreitende Konzentrationswellen, die alle Kennzeichen selbst organisierter Systeme zeigen (Abb. 3). Der Positionierungsapparat funktioniert also offenbar tatsächlich bereits in minimaler Form.
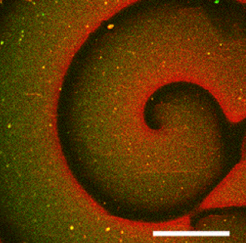
Rekonstitution zellulärer Oszillationen und Vorstadien der Zellteilung
Im nächsten Schritt wurde untersucht, ob die MinDE-Konzentrationswellen in begrenzten Volumina wie Zellen tatsächlich Oszillationen ausbilden und auf diese Weise eine raumzeitliche Positionierung des Teilungsringes erreichen können. Hierzu wurden mithilfe der Mikrosystemtechnik kleine zellähnliche Kompartimente in Silikon erzeugt und mit Membran ausgekleidet. Formt man die Kompartimente in derselben Weise wie eine Bakterienzelle, entstehen bei Zugabe des Teilungsproteins FtsZ tatsächlich nach einiger Zeit allein durch Selbstorganisation der Proteine in der Mitte scharfe Konzentrationsprofile, die der Vorstufe des bakteriellen Teilungsrings entsprechen (Abb. 4).
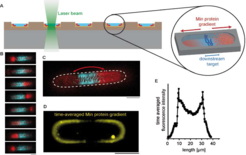
Hiermit sind wichtige Meilensteine hin zu einem Minimalsystem der bakteriellen Zellteilung bereits erreicht, weitere Ziele wie die Rekonstitution dieser Proteinmaschinerie in leicht verformbare Membranvesikel stehen noch aus. Natürlich ist auch ein sich autonom teilendes System noch lange kein Leben, aber doch eine essenzielle Vorstufe, an die sich weitere wichtige Funktionselemente wie die autonome Energieerzeugung (Metabolismus) und vor allem die Replikation einer Informationseinheit (DNA/RNA) anschließen müssen.
Literaturhinweise
Proceedings of the National Academy of Sciences USA 109, 15283-15288 (2012)


![Abb. 3: Selbstorganisationsmuster, die im Reagenzglas durch Rekonstitution der gereinigten Proteine MinD und MinE auf einer künstlichen Membran entstehen [1, 2, 3]. Abb. 3: Selbstorganisationsmuster, die im Reagenzglas durch Rekonstitution der gereinigten Proteine MinD und MinE auf einer künstlichen Membran entste](https://www.mpg.de/11623943/original-1508158007.jpg?t=eyJ3aWR0aCI6ODQ4LCJmaWxlX2V4dGVuc2lvbiI6ImpwZyIsIm9ial9pZCI6MTE2MjM5NDN9--07ce786850ab0fa834ccd1a7bfc55c7110924a00)
![Abb. 4: Rekonstitution einer Teilungszone als Vorstadium des Teilungsrings in minimalen Zellmodellen. (A) zellähnliche Kompartimente in Silikon, die durch Mikrostrukturtechnik hergestellt werden, (B) Gegenoszillationen der Min-Proteine (rot) und der Ringproteine (blau), (C-E) Durch Selbstorganisation eingestellte Konzentrationsprofile mit Vorstufen des Teilungsrings (blau) [4, 5, 6]. Abb. 4: Rekonstitution einer Teilungszone als Vorstadium des Teilungsrings in minimalen Zellmodellen. (A) zellähnliche Kompartimente in Silikon, die d](https://www.mpg.de/11623951/original-1508158007.jpg?t=eyJ3aWR0aCI6ODQ4LCJmaWxlX2V4dGVuc2lvbiI6ImpwZyIsIm9ial9pZCI6MTE2MjM5NTF9--1ea1690635257f672ca992fc27e41eec634886f6)