Die globale Organisation der Chromosomen durch Ringe und Stäbchen
Forschungsbericht (importiert) 2016 - Max-Planck-Institut für Biochemie
Die Organisation von Chromosomen
Jede einzelne Körperzelle benötigt einen vollständigen Satz der genetischen Information, um ihre Funktionen normal erfüllen zu können. Störungen bei der Verteilung der Erbinformation während der Zellteilung führen zu fehlenden oder überschüssigen Erbinformationen, die wiederum die Entwicklung von Krebs fördern können oder Erbdefekte wie zum Beispiel Trisomie 21 bedingen.
Die genetische Information wird in allen Lebewesen in der Abfolge von Nukleobasen in DNA-Molekülen gespeichert. Die Länge der DNA-Moleküle übersteigt dabei die zellulären Dimensionen um mehrere Größenordnungen. Um in der Zelle Platz zu finden und um bei der Zellteilung auf die Tochterzellen aufgeteilt werden zu können, wird die DNA mithilfe von Proteinen dicht gepackt. Die lokale Organisation der DNA übernehmen dabei Histon-Proteine, die kurze DNA-Abschnitte um sogenannte Nukleosomen wickeln. Die Organisation der Chromosomen auf der höheren Ebene blieb aufgrund der enormen Komplexität des Prozesses für lange Zeit weitgehend unbekannt. In den vergangenen Jahren wurden aber neue Methoden entwickelt und optimiert, die jetzt helfen, diese Lücke zu schließen. Bakterien haben sich als hervorragende Modelsysteme etabliert, die meist direkte Rückschlüsse auf verwandte Prozesse in komplexeren Organismen zulassen.
Lokales Laden von SMC – Globale Chromosomen-Faltung
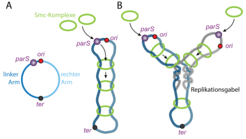
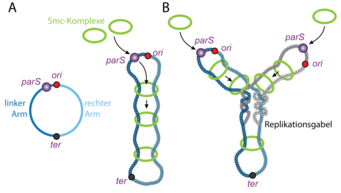
Bakterienzellen besitzen ein einziges, zirkuläres DNA-Molekül, dessen Segmente präzise innerhalb der Zelle angeordnet sind. Für die globale Faltung der DNA ist erstaunlicherweise ein einzelner DNA-Sequenzabschnitt ausschlaggebend. Diese 16 Basen lange Sequenz wird parS genannt. Die Entfernung von parS führt zum Verlust der chromosomalen Organisation, während das Versetzen der parS-Sequenz innerhalb des Chromosoms die globale Organisation auf den Kopf stellt: Die parS-flankierenden DNA-Abschnitte werden dabei jeweils gemäß ihres Abstands von der parS–Sequenz paarweise zusammengeführt, sodass sich zwei gleichgroße Teile des zirkulären Chromosoms der Länge nach zusammenfalten (Abb. 1). Wie aber kann ein einzelner, kleiner DNA-Abschnitt die Organisation des kompletten Chromosoms in seiner Gesamtheit bestimmen?
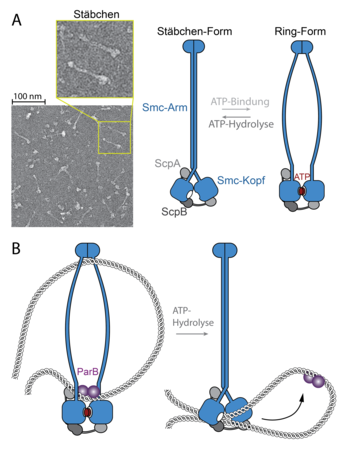
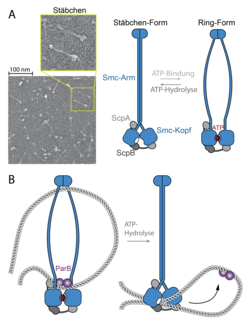
Es stellte sich heraus, dass die parS-Sequenz sich zweier hochkonservierter Systeme für die Organisation des Chromosoms bedient. Das ParB Protein ist Teil des bakteriellen ParABS-Systems. ParB bindet direkt an parS DNA und rekrutiert den zweiten Faktor, den bakteriellen SMC-Komplex, an das Chromosom. Vertreter der Familie der SMC-Proteine spielen eine äußerst wichtige Rolle in der Chromosomenbiologie. Im Gegensatz zu Histonen sind sie auch in Bakterien und Archäen anzutreffen. Gemeinsam mit einer Kleisin-Untereinheit bilden zwei SMC-Proteine eine ringförmige Struktur aus, die in der Lage ist, zwei oder mehrere DNA-Doppelstränge zu umfassen und dadurch in räumlicher Nähe zu halten (Abb. 2). Bis jetzt wurden die SMC-Ringe als passive Klammern für Chromosomen angesehen.
Die Forscher konnten zeigen, dass sich die SMC-Ringe nur kurzfristig in der Nähe der chromosomalen Ladestelle befinden. Unmittelbar nach dem Laden entfernt sich der SMC-Ring entlang der DNA-Doppelhelix von seiner Ladestelle. Die Mobilität der SMC-Ringe auf dem Chromosom ist unerlässlich und steht in direktem Zusammenhang mit Faltung der DNA-Abschnitte, die die Ladestellen links und rechts flankieren.
Molekulare Schmetterlingsflügelschläge
Mit dem Ziel, die molekulare Funktionsweise von SMC-Ringen aufzuklären, studierten die Forscher die Architektur der SMC-Proteine im Detail. Es stellte sich heraus, dass SMC-Komplexe eine hoch-dynamische Struktur aufweisen, die sich durch Bindung und Hydrolyse von ATP dramatisch verändert. Ohne ATP liegt der Komplex nicht in der Form eines offenen Rings vor, sondern nimmt durch die Faltung der langen SMC-Arme eine geschlossene Stäbchen-Form an (Abb. 2). Das SMC-Stäbchen kann weder DNA noch ParB erkennen. Um Kontakt mit der Ladestelle aufzunehmen, muss das SMC-Stäbchen erst mithilfe von zwei ATP-Molekülen, die an die Köpfe der SMC-Proteine binden, aufgebrochen werden (Abb. 2). Bei der Ladestelle angekommen, hydrolysiert der SMC-Komplex beide ATP-Moleküle und verwendet die dabei freigesetzte Energie, um sich von der Ladestelle zu lösen und entlang der flankierenden DNA-Stränge zu wandern. Bei diesem Schritt spielen die Arme der SMC-Proteine wieder eine entscheidende Rolle; dieser Schritt ist allerdings nur oberflächlich aufgeklärt. Möglicherweise arbeiten die SMC-Arme durch die wechselweise Umwandlung in einen Ring und ein Stäbchen als eine Art molekulare Pumpe für DNA-Abschnitte, die DNA-Schleifen schrittweise vergrößert.
Diese Erkenntnisse bilden eine erste Grundlage für das Entschlüsseln verwandter Prozesse in komplexeren Organismen und könnten letztendlich zur Entwicklung von Medikamenten führen, die gezielt in die Funktion der SMC-Komplexe und folglich in die Erhaltung der Erbinformation eingreifen. Neuartige Therapien zur Behandlung von Krebs, Erbkrankheiten oder Antibiotikaresistenzen könnten aus diesen Ergebnissen abgeleitet werden.