Boten-RNA – vermessen und zurechtgestutzt
Forscher des Max-Planck-Instituts für Biochemie entschlüsseln die Struktur eines Ribonukleotid-Verkürzungsmechanismus, der in der Zellbiologie eine zentrale Rolle spielt.
- Boten-RNA (mRNA) überträgt genetische Informationen vom Zellkern an den Ort der Proteinsynthese.
- Die mRNA wird durch eine Kette von Adeninen (Poly(A)-Schwanz) mit Regulierungsfunktionen modifiziert.
- Eine Studie identifiziert die Struktur des Komplexes, der die Verkürzung des Poly(A)-Schwanzes auf die optimale Größe steuert.
- Proteine, die an den Poly(A)-Schwanz binden, messen seine Länge und regulieren parallel dazu die Affinität der den Poly(A)-Schwanz abbauenden Enzyme.
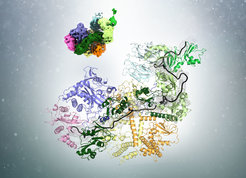
Die Länge des Poly(A)-Schwanzes (schwarz) im Komplex mit dem Poly(A)-Bindeprotein (grün) wird mit Pan2-Pan3 (Blau-, Gelb- und Orange-Töne) gemessen.
Die Boten-RNA (mRNA) stellt das funktionelle Bindeglied zwischen der genetischen Information im Zellkern und den Ribosomen, an denen die Proteinsynthese stattfindet, dar. Die Struktur der mRNA lässt sich in translatierte und nicht translatierte Regionen unterteilen. Die translatierte Region dient als Schablone für die Proteinsynthese, wohingegen die nicht translatierten Regionen Regulierungsfunktionen besitzen. Die nicht translatierten Regionen der mRNA aller höher entwickelten Organismen – angefangen von Hefen bis hin zu Pflanzen und Menschen – weisen ähnliche charakteristische Elemente auf. Ein solches Element ist der Poly(A)-Schwanz: eine lange Kette von Adeninmolekülen, einem der Bausteine der RNA. Diese Schwänze werden nach ihrer Synthese an das Ende der mRNA angehängt und erfüllen zahlreiche Funktionen; dazu gehören die Steuerung der Stabilität, die Translation in Proteine sowie die Lokalisation der mRNA. Naszierende mRNAs besitzen lange Schwänze aus bis zu mehreren hundert Adeninen, die anschließend durch Enzyme, den sogenannten Deadenylasen, auf eine für den jeweiligen Organismus spezifische Länge gekürzt werden.
In einem jüngst in der Fachzeitschrift Cell erschienenen Artikel zeigten Forscher unter der Leitung von Elena Conti am Max-Planck-Institut für Biochemie (MPIB) in Martinsried, wie das Stutzen des Poly(A)-Schwanzes reguliert wird. „Der Poly(A)-Schwanz ist bei seiner Synthese sehr viel länger als später in den Zellen. Daher geht man schon seit langem davon aus, dass es einen Mechanismus zu seiner Verkürzung geben muss. Wie dieser im Detail funktioniert, war allerdings nicht klar“, sagt Ingmar Schäfer, Postdoktorand in Elena Contis Abteilung und Erstautor der Studie. Die Forscher identifizierten jetzt die Struktur der beteiligten Komponenten und zeigten, wie die Messung der Länge und das Stutzen des Poly(A)-Schwanzes miteinander gekoppelt sind. Hierfür ist das Zusammenspiel von drei Komponenten nötig: dem Poly(A)-Schwanz, Poly(A)-Schwanz-bindenden Proteinen (PABP) und dem Pan2-Pan3-Deadenylierungskomplex.
Mit Hilfe der Kryoelektronenmikroskopie (Kryo-EM) fanden die Forscher heraus, dass PABP jeweils etwa 25 bis 30 Basen des Poly(A)-Schwanzes in sich wiederholende Bögen organisiert. Ingmar Schäfer veranschaulicht den Vorgang anhand eines Beispiels aus dem Alltag: „Bevor der Friseur Haare abschneidet, misst er mit den Fingern die gewünschte Haarlänge.“ In ähnlicher Weise dienen die PABP-Bögen zur Messung der Länge des Poly(A)-Schwanzes. „Anders als unsere Haare beim Friseur, wird der Poly(A)-Schwanz aber nicht mit einem Schnitt gekürzt, sondern vielmehr von hinten durch die Deadenylase ‚abgeknabbert‘.“
Interessanterweise verändert die Anzahl der gebundenen PABP-Moleküle die Affinität der Deadenylase für den Poly(A)-Schwanz. Bei Hefe beträgt die optimale Länge des Poly(A)-Schwanzes etwa 30 Nukleotide. An längere Poly(A)-Schwänze können mehrere PABP gebunden sein, so dass die Adeninkette rasch abgebaut wird. Kürzere Poly(A)-Schwänze mit fast optimaler Länge können lediglich einen Messproteinkomplex binden, was einer geringeren Affinität für die Deadenylasen entspricht. „Daher dient PABP nicht nur als Instrument zur Messung des Poly(A)-Schwanzes, sondern auch als Bremse für die Deadenylasen, sobald die optimale Länge erreicht ist“, erklärt Schäfer. Schließlich wird die mRNA, wenn sie nicht länger benötigt wird, durch andere Deadenylasen weiter abgebaut.
Die Polyadenylierung ist ein Grundprinzip der Zellbiologie und dient der Steuerung der mRNA-Stabilität und Proteinsynthese. „Die Struktur dieses ‚Stutzwerkzeugs‘ für den Poly(A)-Schwanz wird Auswirkungen auf die Erforschung verschiedenster Aspekte der Zellbiologie haben“, prophezeit MPIB-Direktorin Elena Conti und stellt die Studie damit in einen größeren Zusammenhang. Der Mechanismus, so Conti, sei von Hefen bis hin zum Menschen stark konserviert. „Jetzt wo wir die Struktur der Deadenylierungsmaschinerie bei Hefe entschlüsselt haben, möchten wir verstehen, wie das System in menschlichen Zellen funktioniert, in denen die optimale Länge des Poly(A)-Schwanzes eine andere ist als bei Hefe.“
Originalpublikation:
I.B. Schäfer, M. Yamashita, J.M. Schuller, S. Schüssler, P. Reichelt, M. Strauss, E. Conti: Molecular basis for poly(A) RNP architecture and recognition by the Pan2-Pan3 deadenylase. Cell, Mai 2019
DOI: https://doi.org/10.1016/j.cell.2019.04.013












